
핵산정량(Nucleic Acid Quant ification)
핵산 정량이란 샘플에서 추출한 DNA, RNA의 양(농도)을 확인하는 과정입니다.가장 일반적으로 사용되는 샘플에는 동물/세포조직, 혈액, 구강표본, 체액, 진핵세포와 박테리아 등이 있으며, 이렇게 추출된 핵산은 다양한 실험에 사용됩니다.
핵산(Nucleic Acid)이란 무엇일까요?
핵산(Nucleic Acid) 핵속에 들어있는 산성물질이라는 의미로 핵산이라 이름붙여 뉴클레오티드(Nucleotides)라는 단위체로 구성된 중합체로 유전정보의 보존과 전달, 발협을 돕는 기능을 하며 푸딩, 피리미딘 염기의 공명구조를 통해 260nm로부터의 파장을 강하게 흡수합니다.
뉴클레오티드: 1개의 오탄당(Pentose)과 1개의 염기(base)와 1개 이상의 인산(Phosphate)이 합쳐진 구조 오탄당 탄소원자 5개로 이루어진 단당류 염기푸딩계(환구조 2개)-아데닌(Adenine), 구아닌(Guanine) 피리미딘계(환구조 1개)-사이토신(Cyt).
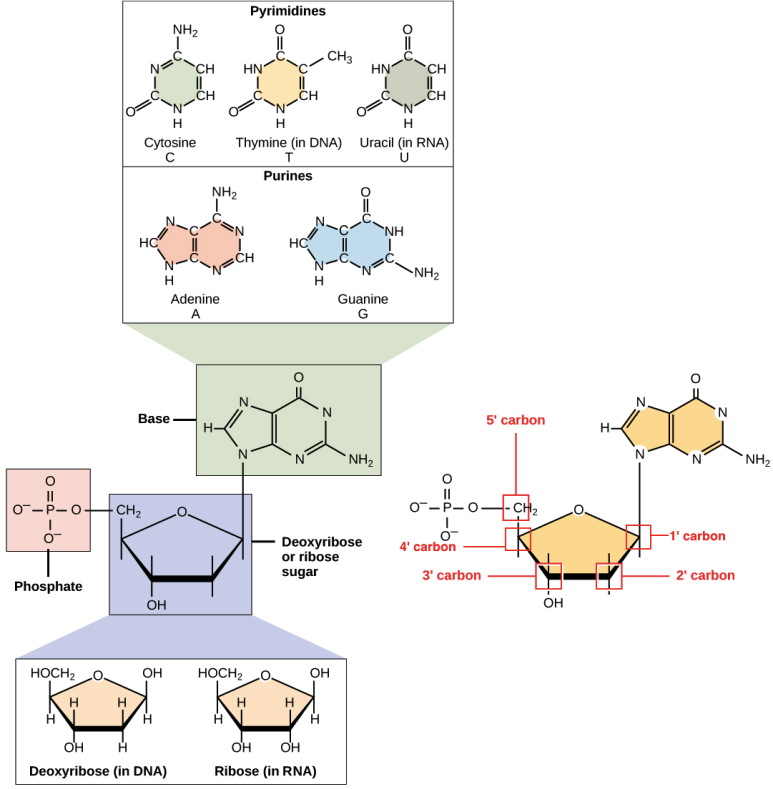
DNA(Deoxyribonucleic Acid) 뉴클레오타이드의 중합체인 두 개의 긴 모서리가 서로 뒤틀리는 이중 나선 구조로 된 고분자 화합물
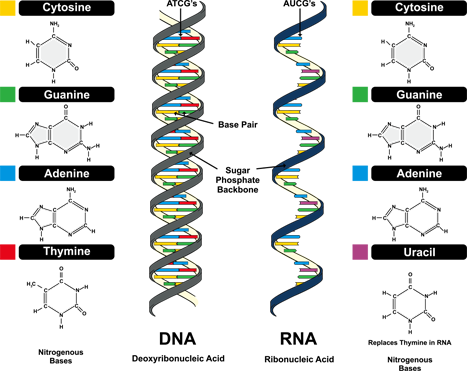
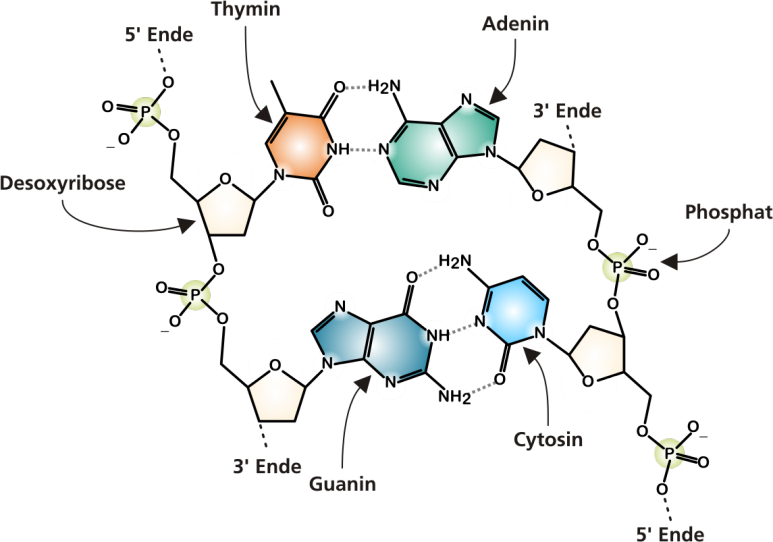
외사슬(Singlestranded) DNA(ss DNA)는 디옥시리보스의 3’탄소가 다음 디옥시리보스의 5’탄소와 Phosphodiester를 결합하여 5’>3′ 방향으로 연결됩니다. 이 ssDNA는 반대편의 염기와 수소결합으로 두 가닥(Doublestranded) DNA(dsDNA)를 형성하여 결국 오른쪽 방향으로 꼬인 이중나선구조가 됩니다.dsDNA 사이에서 염기 결합은 A와 T가 이중수소 결합을 하고 G와 C가 삼중수소 결합으로 쌍을 이루는데 이를 상보적 결합(Complementary)이라고 합니다.
수소결합 개수로 인해 A-T결합은 G-C결합에 비해 결합력이 약하여 상대적으로 파손되기 쉽고 DNA손상 시 결합력이 약한 A-T결합이 우선적으로 파괴되는 경향이 있습니다.
RNA(Ribo Nucleic Acid)와 당다리 분자에 인산과 염기가 붙은 외가닥 모양의 구조로 된 고분자
RNA의 구조는 3개의 측면에서 DNA와 차이를 가지고 있습니다.”첫째는 5탄당 2’탄소에 수산화(OH)기가 부착된 리보오스(Ribose)이고, 둘째는 염기가 티민(Thymine) 대신 우라실(Uracil)이고, 셋째는 1가지(Single-stranded)입니다”RNA는 DNA와는 다른 한줄기 구조로 리보스의 2′-OH가 알칼리 가수분해로 인해 쉽게 분해되므로 DNA에 비해 불안정하고(DNA에 비해 결합력이 떨어진다) 일상환경에 존재하는 RNase(RNA를 분해하는 효소)에 의해 파괴되기 쉬워집니다.
DNA추출방법(DNA Prep)
DNA 정량 앞에 DNA를 추출하는 과정이 선행되어야 하는데 이에 대해 살펴보겠습니다.
세포에서 DNA를 분리하려면 우선 세포 내 DNA가 세포 밖으로 유출되도록 세포벽 및 세포막을 분해해야 한다.
세포벽 세포를 외부로부터 보호하고 세포의 형태를 유지할 수 있도록 하는 벽을 말합니다.원핵세포의 세균, 진핵세포의 식물세포나 곰팡이, 고균 등이 세포벽을 가지고 있으며 셀룰로오스, 펙틴, 리그닌, 큐틴 등이 주성분입니다.
세포막세포와 세포외부의 경계를 이루는 막으로 세포내 물질을 보호하고 세포간 물질의 이동을 조절합니다.포막의 주요 구성성분은 양친매성(amphipathic)을 나타내는 지질로서 막 내부는 소수성(hydrophobic, 비극성)이며 외부는 친수성(hydrophilic, 극성)의 이중층으로 이루어져 있습니다.지질,단백질,탄수화물이주로포함됨.
세포벽과 세포막을 파괴하는 방법은 물리적인 방법과 화학적인 방법으로 나뉘는데 물리적 방법은 기계적인 힘으로 세포를 파괴하는 것이고 화학적 방법은 화학약품을 통해 세포벽을 용해시키는 것을 의미한다.DNA 분리를 위해서는 주로 화학적 방법이 사용된다.
세포막의 주성분은 부모의 매실성을 나타내는 지질로 구성되어 있는데 계면활성제 종류인 Sodium Dodecyl Sulfate(SDS)를 통해 세포막을 분해하여 DNA를 추출합니다.이렇게 되면 세포는 죽고 세포 내 물질이 버퍼 등의 수용액에 떠 버립니다.이 과정을 lysis 과정이라고 합니다.
이렇게 추출된 DNA에는 히스톤 단백질이 함유되어 있는데 이러한 단백질을 제거해 주는 용액 Proteinase K(단백질을 가수분해하는 효소)를 사용하여 히스톤 단백질을 제거해야 합니다.히스톤 단백질은 DNA의 안정성을 유지하는 역할을 합니다.그러나 실험을 위해 DNA를 추출하는 과정에서 히스톤 단백질이 부착되어 있으면 DNA의 Purity(순도)가 좋지 않으므로 이를 제거합니다.
다음으로 페놀(Phenol)/클로로포름(Chlor oform)/is oamyl alcohol(25:24:1)이 첨가되어 원심분리가 진행되면 Phenol 성분이 단백질과 결합하여 단백질을 침전시킵니다.버퍼 안에는 수많은 단백질이 존재하지만 이를 침전시키지 않으면 순수한 DNA를 얻기 어렵기 때문에 이러한 과정에서 단백질을 침전시켜주는 것입니다.
이렇게 단백질을 침전시킨 후 단백질을 침전시키는 역할을 했던 Phenol을 없애야 합니다.Phenol은 단백질을 변성시키고 실험에도 좋지 않은 결과를 미칠 수 있으므로 이를 클로로포름(Chlor oform)을 통해 DNA 수용층과 Phenol 수용층을 분리하여 상단에 DNA 수용층이 있는 부분만을 잘 추출하여 실험에 사용할 수 있도록 합니다.is oamyl alcohol을 넣는 이유는 Phenol과 클로로포름이 섞이도록 하기 위해서인데 해당 물질이 없는 경우 사용하지 않아도 됩니다.
아이소프로판올(is opropanol)이나 에탄올(ethanol)은 DNA의 charge에 의해 물에 쉽게 녹기 때문에 침전이 원활하지 않을 때 사용하는 물질입니다.uncharge 물질이기 때문에 이로 인해 DNA의 물에 대한 친화력을 떨어뜨려 원심 분리를 할 때 잘 침전되도록 하는 것입니다.
DNA를 추출하는 방법과 RNA를 추출하는 방법에는 차이가 있으나 RNA는 DNA와 달리 산성에 강하고 알칼리성에 약합니다.보통 Trizol이라는 물질을 이용하는데, guanidine is othiocyanate가 RNase 억제를 Phenol은 단백질과 지질을 붕괴시키고 혼합물에 클로로포름을 넣어 수성층과 유기층을 분리시킨 후 Isopropanol을 통해 RNA를 침전시킵니다.이어서 RNATE Buffer 또는 Rnase Freewater로 용해하여 RNA를 수행합니다.
장점으로는 RNA가 변성이 없고 RNA회수율과 순도가 높지만 유해물질 성분이기 때문에 사용상 주의를 요하는 방법입니다.
DNA, RNA 정량방법(DNAquantification)
이렇게 순차적으로 DNA 또는 RNA 추출까지 이루어지면 향후 계속될 실험에 적당한 조건을 갖추고 있는지를 확인하기 위해 핵산 정량이라는 과정을 거치게 됩니다. 이를 통해 핵산의 양(Yield)과 순도(Purity)를 확인합니다.
용액 속의 DNA 양을 측정하기 위해서는 두 가지 방법이 있습니다.하나는 일반적인 Spectrophotometer를 이용하는 방법과 하나는 Nano Spectrophotometer를 이용하는 방법입니다.
일반적인 UV-VIS Spectrophotometer의 경우 시료의 양이 어느정도 확보되었을때 사용할 수 있어 정확도 및 재현성이 좋은편입니다.단점으로는 시료의 양이 너무 적은 경우에는 사용이 불가하고 다소 실험과정에 Step이 추가된다는 점이 있습니다.
Nano Spectrophotometer의 경우 1ul정도의 극미량의 시료로도 측정이 가능해 간편하다는 장점이 있지만 기기가 일반적인 UV-VIS Spectrophotometer보다 높아 해당 작업을 진행하는 작업자의 수준에 따라 정밀도 및 재현성에 차이가 있는 편입니다.
DNA의 농도는 260nm에서 흡광도(obtical density)를 측정하여 확인합니다.
핵산의 정량 계산 수식은 이렇습니다.[OD260nmXDNA or RNA 상수X희석배수=ug/ml] OD값이 1일 때 그 안에 들어있는 핵산의 농도(ug/ml 단위)를 정수와 같이 사용하여 계산에 사용합니다.
이중본 DNA(Doublestranded DNA, ds DNA) 260nm에서 흡광도(Absorbance) 값이 1일 경우 50ug/ml
외사슬 DNA(Singlestranded DNA, ss DNA) 260nm에서 흡광도(Absorbance) 값이 1이면 33ug/ml
RNA값을 DNA에 비해 낮게 설정하는 이유는 RNA의 흡광도가 높기 때문입니다.
예를 들어 DNA 추출시료를 녹인 용매 100μl 중 10μl를 취하며 이를 100배 희석한 후 OD 값이 0.400이 나왔다면 시료 10μl 중 DNA 총량이 몇 ug인지는 다음과 같이 계산할 수 있다.
[OD260nmXDNA 상수X희석배수=ug/ml]
0.400*50ug/ml*10=2000ug/ml라는 값을 얻는데 이는 시료 1ml당 2000ug의 DNA가 들어있음을 의미한다.그런데 계산을 요하는 시료의 양은 10μl=0.01ml이므로 2000ug/ml*0.01ml를 하면 시료 10μl 중 DNA 총량은 20ug이다.DNA순도(DNA Purity)
DNA의 순도(Purity)는 260nm의 흡광도 값을 280nm 또는 230nm 값의 비율로 정합니다.
A260/280rio값에 의한 DNA순도측정 DNA의 경우 A260/280 값이 1.8~1.9인 경우: 순도가 좋다.
A260/280rio 값이 낮은 경우-DNA 농도가 낮은RNA(Ribo Nucleic Acid)와 당다리 분자에 인산과 염기가 붙은 외가닥 모양의 구조로 된 고분자
RNA의 구조는 3개의 측면에서 DNA와 차이를 가지고 있습니다.”첫째는 5탄당 2’탄소에 수산화(OH)기가 부착된 리보오스(Ribose)이고, 둘째는 염기가 티민(Thymine) 대신 우라실(Uracil)이고, 셋째는 1가지(Single-stranded)입니다”RNA는 DNA와는 다른 한줄기 구조로 리보스의 2′-OH가 알칼리 가수분해로 인해 쉽게 분해되므로 DNA에 비해 불안정하고(DNA에 비해 결합력이 떨어진다) 일상환경에 존재하는 RNase(RNA를 분해하는 효소)에 의해 파괴되기 쉬워집니다.
DNA추출방법(DNA Prep)
DNA 정량 앞에 DNA를 추출하는 과정이 선행되어야 하는데 이에 대해 살펴보겠습니다.
세포에서 DNA를 분리하려면 우선 세포 내 DNA가 세포 밖으로 유출되도록 세포벽 및 세포막을 분해해야 한다.
세포벽 세포를 외부로부터 보호하고 세포의 형태를 유지할 수 있도록 하는 벽을 말합니다.원핵세포의 세균, 진핵세포의 식물세포나 곰팡이, 고균 등이 세포벽을 가지고 있으며 셀룰로오스, 펙틴, 리그닌, 큐틴 등이 주성분입니다.
세포막세포와 세포외부의 경계를 이루는 막으로 세포내 물질을 보호하고 세포간 물질의 이동을 조절합니다.포막의 주요 구성성분은 양친매성(amphipathic)을 나타내는 지질로서 막 내부는 소수성(hydrophobic, 비극성)이며 외부는 친수성(hydrophilic, 극성)의 이중층으로 이루어져 있습니다.지질,단백질,탄수화물이주로포함됨.
세포벽과 세포막을 파괴하는 방법은 물리적인 방법과 화학적인 방법으로 나뉘는데 물리적 방법은 기계적인 힘으로 세포를 파괴하는 것이고 화학적 방법은 화학약품을 통해 세포벽을 용해시키는 것을 의미한다.DNA 분리를 위해서는 주로 화학적 방법이 사용된다.
세포막의 주성분은 부모의 매실성을 나타내는 지질로 구성되어 있는데 계면활성제 종류인 Sodium Dodecyl Sulfate(SDS)를 통해 세포막을 분해하여 DNA를 추출합니다.이렇게 되면 세포는 죽고 세포 내 물질이 버퍼 등의 수용액에 떠 버립니다.이 과정을 lysis 과정이라고 합니다.
이렇게 추출된 DNA에는 히스톤 단백질이 함유되어 있는데 이러한 단백질을 제거해 주는 용액 Proteinase K(단백질을 가수분해하는 효소)를 사용하여 히스톤 단백질을 제거해야 합니다.히스톤 단백질은 DNA의 안정성을 유지하는 역할을 합니다.그러나 실험을 위해 DNA를 추출하는 과정에서 히스톤 단백질이 부착되어 있으면 DNA의 Purity(순도)가 좋지 않으므로 이를 제거합니다.
다음으로 페놀(Phenol)/클로로포름(Chlor oform)/is oamyl alcohol(25:24:1)이 첨가되어 원심분리가 진행되면 Phenol 성분이 단백질과 결합하여 단백질을 침전시킵니다.버퍼 안에는 수많은 단백질이 존재하지만 이를 침전시키지 않으면 순수한 DNA를 얻기 어렵기 때문에 이러한 과정에서 단백질을 침전시켜주는 것입니다.
이렇게 단백질을 침전시킨 후 단백질을 침전시키는 역할을 했던 Phenol을 없애야 합니다.Phenol은 단백질을 변성시키고 실험에도 좋지 않은 결과를 미칠 수 있으므로 이를 클로로포름(Chlor oform)을 통해 DNA 수용층과 Phenol 수용층을 분리하여 상단에 DNA 수용층이 있는 부분만을 잘 추출하여 실험에 사용할 수 있도록 합니다.is oamyl alcohol을 넣는 이유는 Phenol과 클로로포름이 섞이도록 하기 위해서인데 해당 물질이 없는 경우 사용하지 않아도 됩니다.
아이소프로판올(is opropanol)이나 에탄올(ethanol)은 DNA의 charge에 의해 물에 쉽게 녹기 때문에 침전이 원활하지 않을 때 사용하는 물질입니다.uncharge 물질이기 때문에 이로 인해 DNA의 물에 대한 친화력을 떨어뜨려 원심 분리를 할 때 잘 침전되도록 하는 것입니다.
DNA를 추출하는 방법과 RNA를 추출하는 방법에는 차이가 있으나 RNA는 DNA와 달리 산성에 강하고 알칼리성에 약합니다.보통 Trizol이라는 물질을 이용하는데, guanidine is othiocyanate가 RNase 억제를 Phenol은 단백질과 지질을 붕괴시키고 혼합물에 클로로포름을 넣어 수성층과 유기층을 분리시킨 후 Isopropanol을 통해 RNA를 침전시킵니다.이어서 RNATE Buffer 또는 Rnase Freewater로 용해하여 RNA를 수행합니다.
장점으로는 RNA가 변성이 없고 RNA회수율과 순도가 높지만 유해물질 성분이기 때문에 사용상 주의를 요하는 방법입니다.
DNA, RNA 정량방법(DNAquantification)
이렇게 순차적으로 DNA 또는 RNA 추출까지 이루어지면 향후 계속될 실험에 적당한 조건을 갖추고 있는지를 확인하기 위해 핵산 정량이라는 과정을 거치게 됩니다. 이를 통해 핵산의 양(Yield)과 순도(Purity)를 확인합니다.
용액 속의 DNA 양을 측정하기 위해서는 두 가지 방법이 있습니다.하나는 일반적인 Spectrophotometer를 이용하는 방법과 하나는 Nano Spectrophotometer를 이용하는 방법입니다.
일반적인 UV-VIS Spectrophotometer의 경우 시료의 양이 어느정도 확보되었을때 사용할 수 있어 정확도 및 재현성이 좋은편입니다.단점으로는 시료의 양이 너무 적은 경우에는 사용이 불가하고 다소 실험과정에 Step이 추가된다는 점이 있습니다.
Nano Spectrophotometer의 경우 1ul정도의 극미량의 시료로도 측정이 가능해 간편하다는 장점이 있지만 기기가 일반적인 UV-VIS Spectrophotometer보다 높아 해당 작업을 진행하는 작업자의 수준에 따라 정밀도 및 재현성에 차이가 있는 편입니다.
DNA의 농도는 260nm에서 흡광도(obtical density)를 측정하여 확인합니다.
핵산의 정량 계산 수식은 이렇습니다.[OD260nmXDNA or RNA 상수X희석배수=ug/ml] OD값이 1일 때 그 안에 들어있는 핵산의 농도(ug/ml 단위)를 정수와 같이 사용하여 계산에 사용합니다.
이중본 DNA(Doublestranded DNA, ds DNA) 260nm에서 흡광도(Absorbance) 값이 1일 경우 50ug/ml
외사슬 DNA(Singlestranded DNA, ss DNA) 260nm에서 흡광도(Absorbance) 값이 1이면 33ug/ml
RNA값을 DNA에 비해 낮게 설정하는 이유는 RNA의 흡광도가 높기 때문입니다.
예를 들어 DNA 추출시료를 녹인 용매 100μl 중 10μl를 취하며 이를 100배 희석한 후 OD 값이 0.400이 나왔다면 시료 10μl 중 DNA 총량이 몇 ug인지는 다음과 같이 계산할 수 있다.
[OD260nmXDNA 상수X희석배수=ug/ml]
0.400*50ug/ml*10=2000ug/ml라는 값을 얻는데 이는 시료 1ml당 2000ug의 DNA가 들어있음을 의미한다.그런데 계산을 요하는 시료의 양은 10μl=0.01ml이므로 2000ug/ml*0.01ml를 하면 시료 10μl 중 DNA 총량은 20ug이다.DNA순도(DNA Purity)
DNA의 순도(Purity)는 260nm의 흡광도 값을 280nm 또는 230nm 값의 비율로 정합니다.
A260/280rio값에 의한 DNA순도측정 DNA의 경우 A260/280 값이 1.8~1.9인 경우: 순도가 좋다.
A260/280rio 값이 낮은 경우-DNA 농도가 낮은 경우
- DNA 농도는 정상이나 ratio 값이 낮은 경우-단백질 오염도가 높음-DNA가 녹아 있는 용액의 PH가 낮은 경우
- A260/230 값이 높은 경우 – DNA 농도가 높은 경우 – 값이 2.0 이상인 경우 RNA Contamination(오염), DNA Fragmentaion(DNA 분열)
- A260/230 ratio 값에 의한 순도 측정 DNA의 경우 A260/230 값이 2.0~2.2인 경우: 순도가 좋다.
- A260/230 ratio 값이 낮은 경우 – contaminant(불순물)에 의해 값이 낮게 나오는 – Cabohydrate(식물에서 DNA 추출한 경우) – Phenol을 사용하는 DNA 추출 과정에서 Phenol이 남아 있는 경우 – Guanidine(spincolumn 방식의 Prepkit에서 DNA 추출 buffer 성분)이 남아 있는 경우 – DNA 추출 과정에서 Prepkit
- 이상 DNA, RNA 정량에 대해서 조사하였습니다.
- DNA정량추가를위한Nano Spectrophotometer제품추천
- 경우
- DNA 농도는 정상이나 ratio 값이 낮은 경우-단백질 오염도가 높음-DNA가 녹아 있는 용액의 PH가 낮은 경우
- A260/230 값이 높은 경우 – DNA 농도가 높은 경우 – 값이 2.0 이상인 경우 RNA Contamination(오염), DNA Fragmentaion(DNA 분열)
- A260/230 ratio 값에 의한 순도 측정 DNA의 경우 A260/230 값이 2.0~2.2인 경우: 순도가 좋다.
- A260/230 ratio 값이 낮은 경우 – contaminant(불순물)에 의해 값이 낮게 나오는 – Cabohydrate(식물에서 DNA 추출한 경우) – Phenol을 사용하는 DNA 추출 과정에서 Phenol이 남아 있는 경우 – Guanidine(spincolumn 방식의 Prepkit에서 DNA 추출 buffer 성분)이 남아 있는 경우 – DNA 추출 과정에서 Prepkit
- 이상 DNA, RNA 정량에 대해서 조사하였습니다.
- DNA정량추가를위한Nano Spectrophotometer제품추천

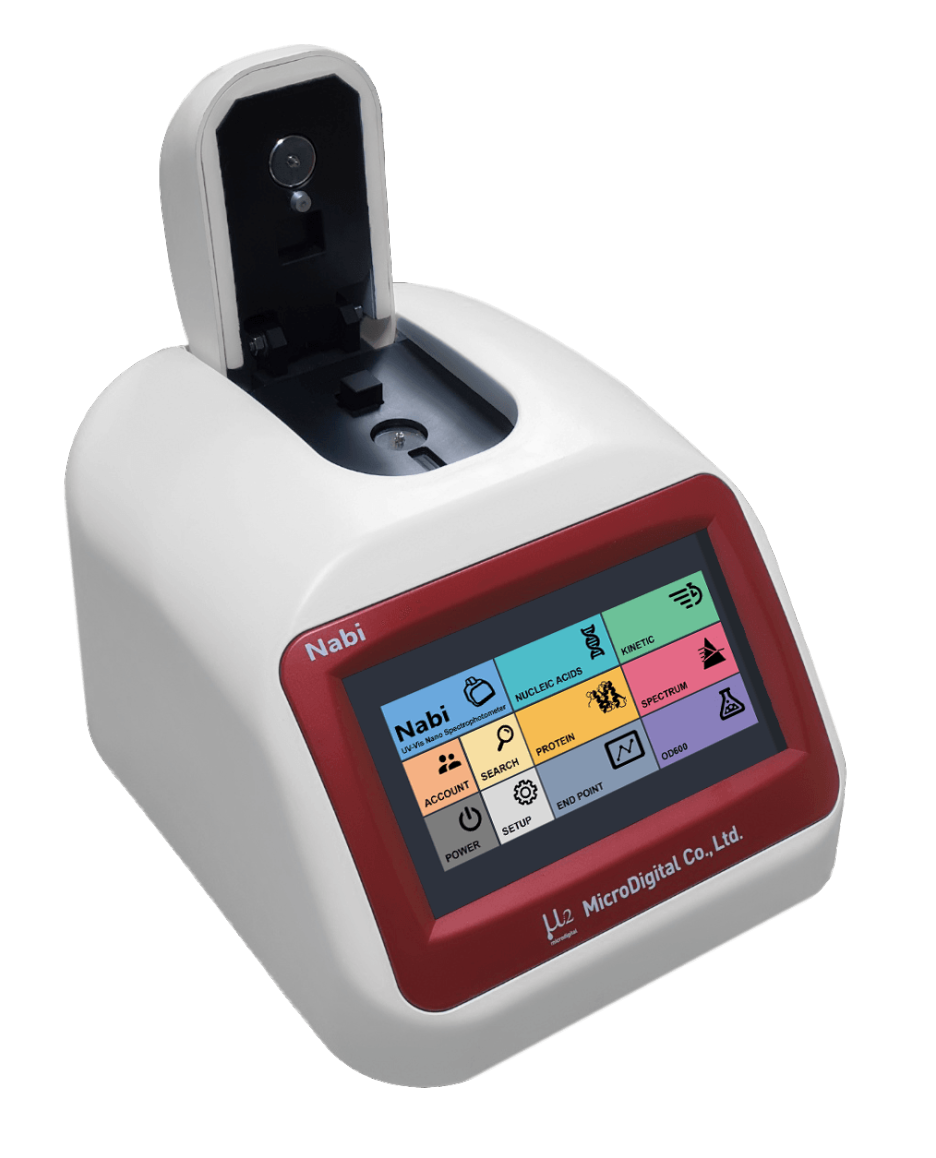
(주)마이크로디지털 NanoS pectrophotometer Nabi